MinION : le nouveau mini-séquenceur portatif du GEVES
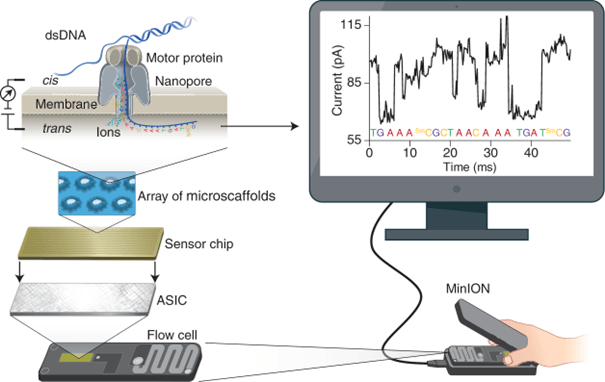
Le GEVES à Beaucouzé s’est doté d’un séquenceur haut débit : le MinION Mk1B. Ce séquenceur de 3ème génération qui tient dans une main permet de réaliser du séquençage en temps réel de longs fragments d’ADN ou d’ARN.
A la suite de cette acquisition et avec les conseils précieux de Jessica Dittmer (INRAE – IRHS), le GEVES a séquencé avec succès sa première souche bactérienne : la souche de référence de Xanthomonas campestris pv. raphani 756C. En effet, le génome nouvellement séquencé présente 99.91% d’identité avec le génome disponible dans les bases de données publiques. Ainsi, le GEVES va notamment pouvoir caractériser la collection d’agents pathogènes qu’il possède au sein du laboratoire de pathologie du GEVES.
De plus, plusieurs axes de développement sont envisagés pour intégrer le MinION dans les missions du pôle Détection du GEVES, comme l’identification de variation génétique, le multiplexage d’échantillons, la détection d’OGM ou la détection de multiples agents pathogènes dans un échantillon de semences en une seule analyse par métabarcoding.
Pour ce dernier axe de développement, un projet CASDAR coordonné par le GEVES, en partenariat avec INRAE, la SFR QuaSaV-ANAN et des semenciers (HM.Clause, Vilmorin-Mikado et Sakata Europe), envisage d’expérimenter cette méthode de détection : le projet SeqDetectVeg.